1. 細胞抽出ページ
トップページからCell Extractionを選択します。
ブラウザからND2ファイルをアップロードしてボタンをクリックするだけで、
画像に含まれる細胞の輪郭情報を自動抽出、SQLiteデータベースに格納します。
実験系ごとにデータベースを作成し、ブラウザ上で1細胞ごとの蛍光強度や位置情報などの各種統計量を確認、
さらに機械学習用に任意の教師ラベルを付与できます。
細胞データベースから1細胞ごとの各種統計量をCSV形式で出力し、
集団レベルの情報をブラウザ上で可視化。
複数人が同時にブラウザからアクセスして、同じ細胞データベースに対してラベリングを行うことができます。
チームの力で教師データを高速に作成できます。
細胞データベースに接続して、任意の実験系における任意の細胞の情報を瞬時に参照。

特定の細胞集団における、各細胞の細胞内GFP蛍光強度をヒートマップで可視化。

位相差画像と輪郭のペアから、洗練された教師データを簡単に作成。
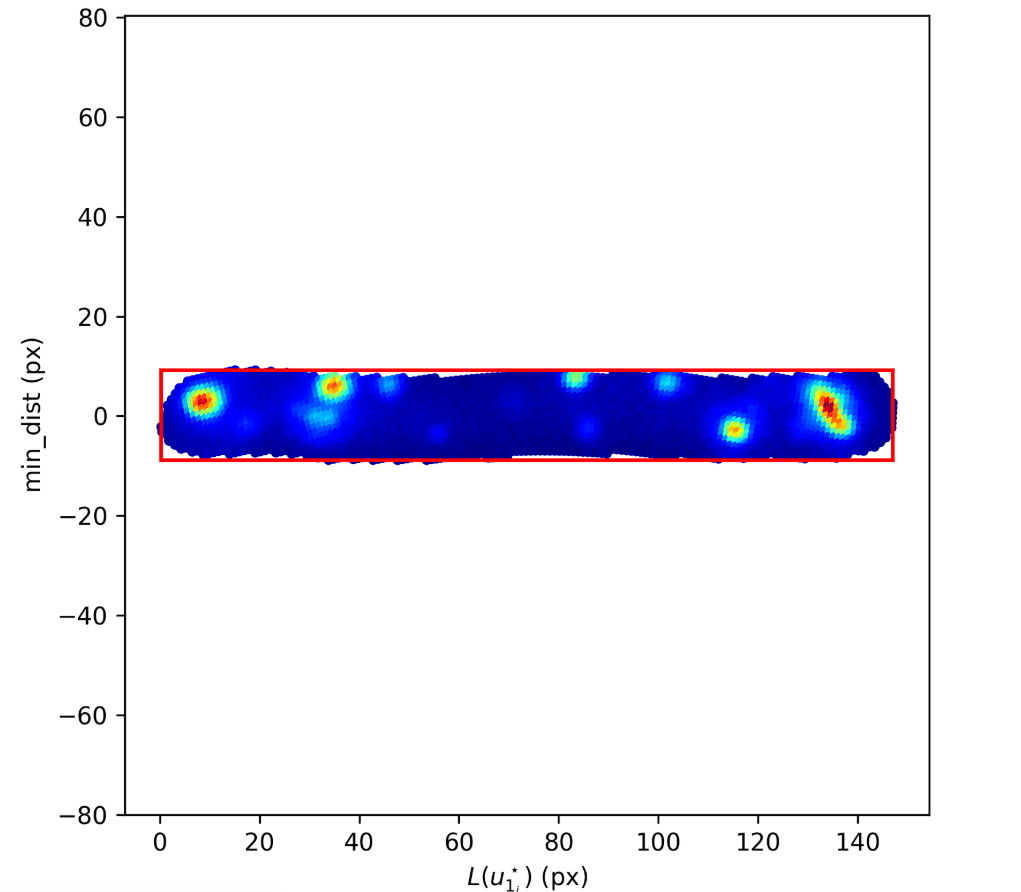
湾曲した細胞を直線化して、細胞内の蛍光強度分布を解析するなどの、任意のアルゴリズムを組み込み可能。
PhenoPixel5.0はローカルネットワークのサーバー上で動作させることを想定して設計されています。
下記の手順に沿って操作すると、データの取り込みから解析・出力までスムーズに行えます。各ステップのスクリーンショットをご参照ください。

トップページからCell Extractionを選択します。
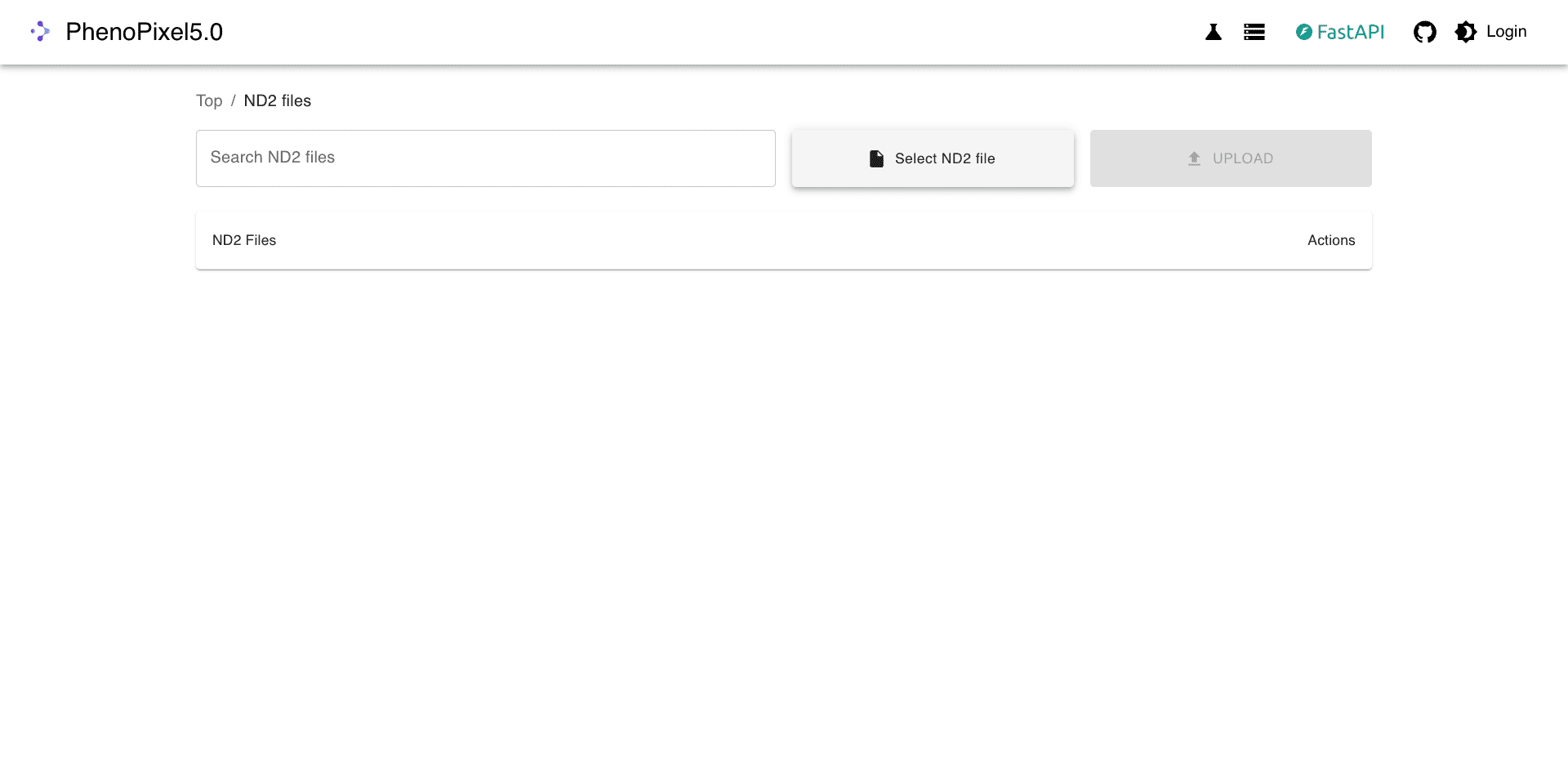
Select ND2をクリックして、ND2ファイルをアップロードします。
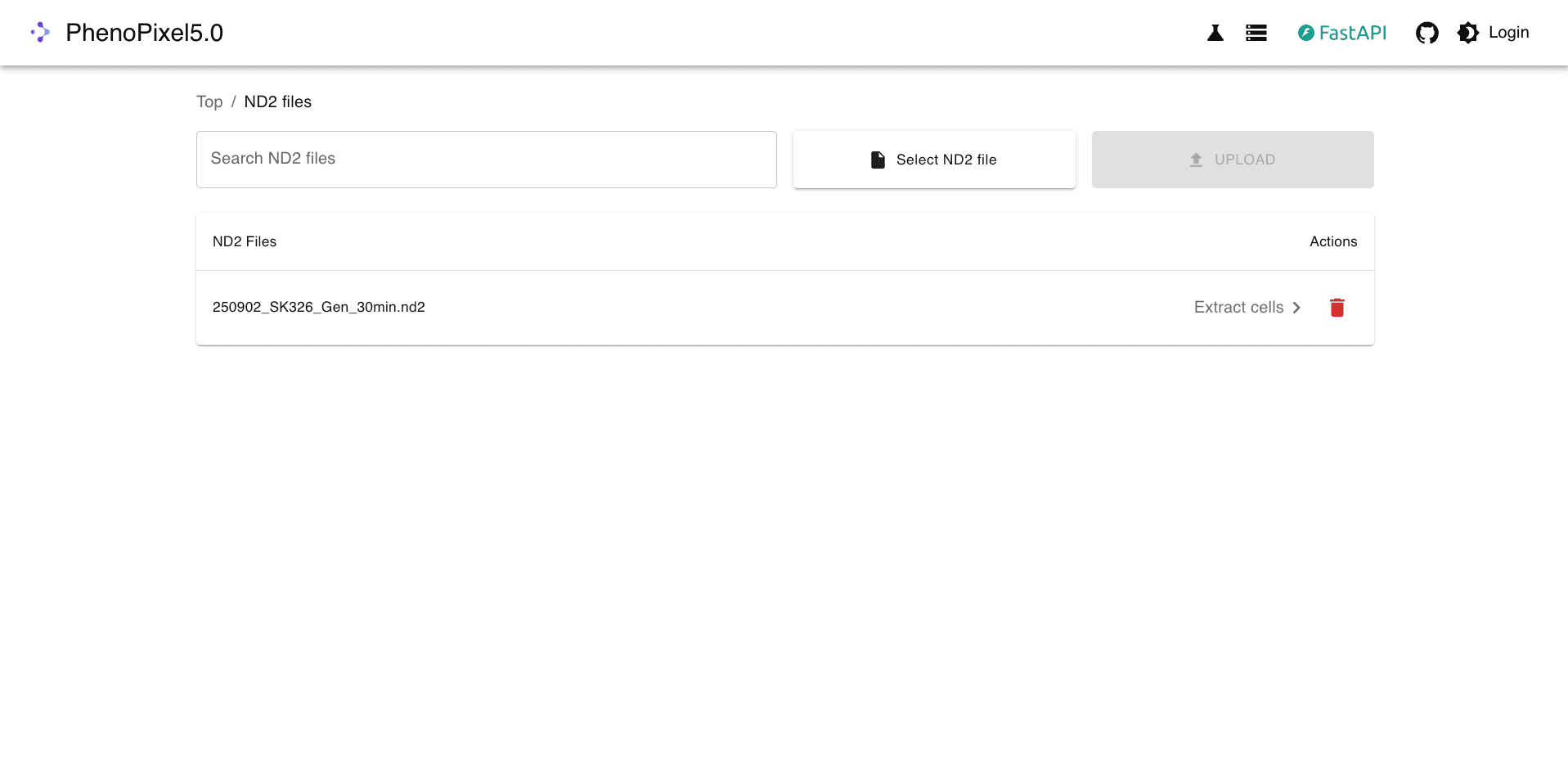
解析対象のファイルについて、Extract Cellsをクリックします。

画像に含まれる細胞の輪郭情報を自動抽出し、SQLiteデータベースに格納します。Modeはチャネル数に合わせてDualまたはTripleを選択します。Param1はCannyアルゴリズムの内部パラメータに相当し、デフォルトの100を使用しますがPHチャネルが暗い場合は75などに調整します。Image Sizeは1細胞あたりの単位画像の辺サイズ(px)で、細胞サイズに応じて設定します。パラメータの設定後、Extract Cellsをクリックします。
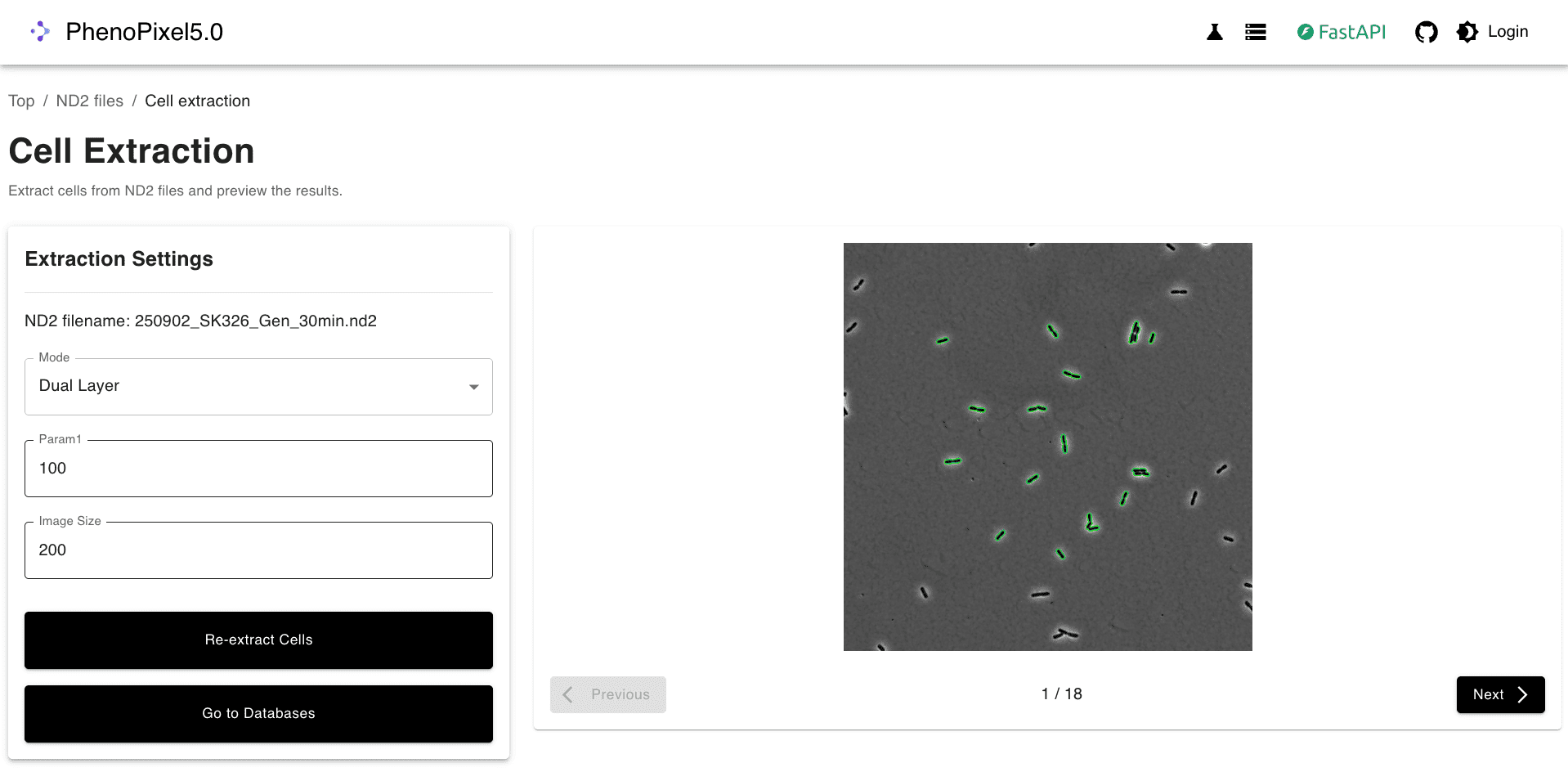
輪郭が正常に検出できているかを確認します。必要に応じてパラメータを調整し、再度Extract Cellsをクリックします。画面端で検出が弱い場合はパラメータを再調整してください。NEXTおよびPreviousで全視野を確認し、問題がなければGo to DatabaseでSQLiteデータベースに保存します。
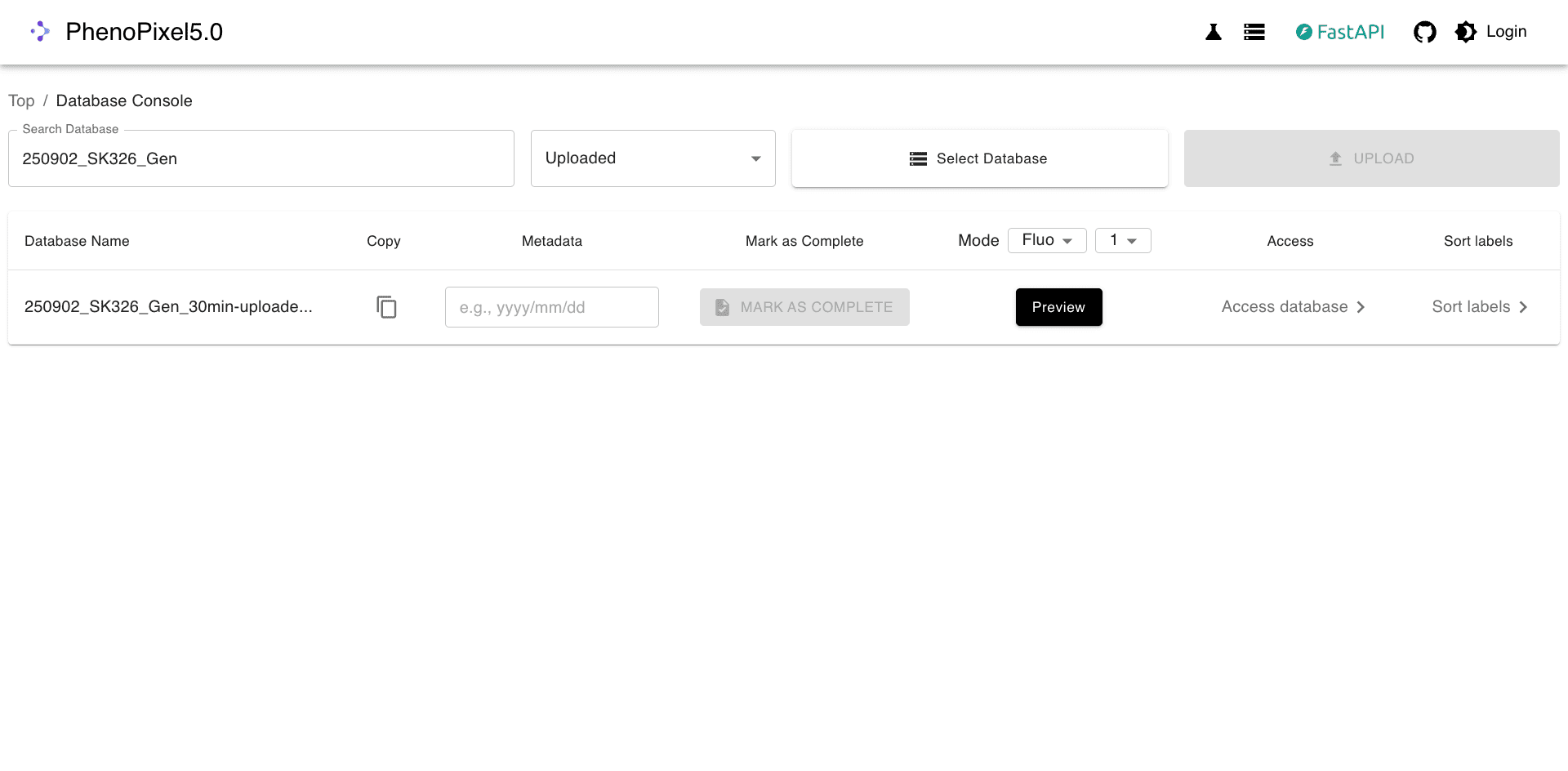
Database Consoleに前ステップで作成したSQLiteデータベースが表示されます。Access databaseをクリックし、1細胞ごとに蛍光強度や位置情報などの統計量を確認します。Sort Labelsを選択すると、UI上で直感的にラベリングを行えます。
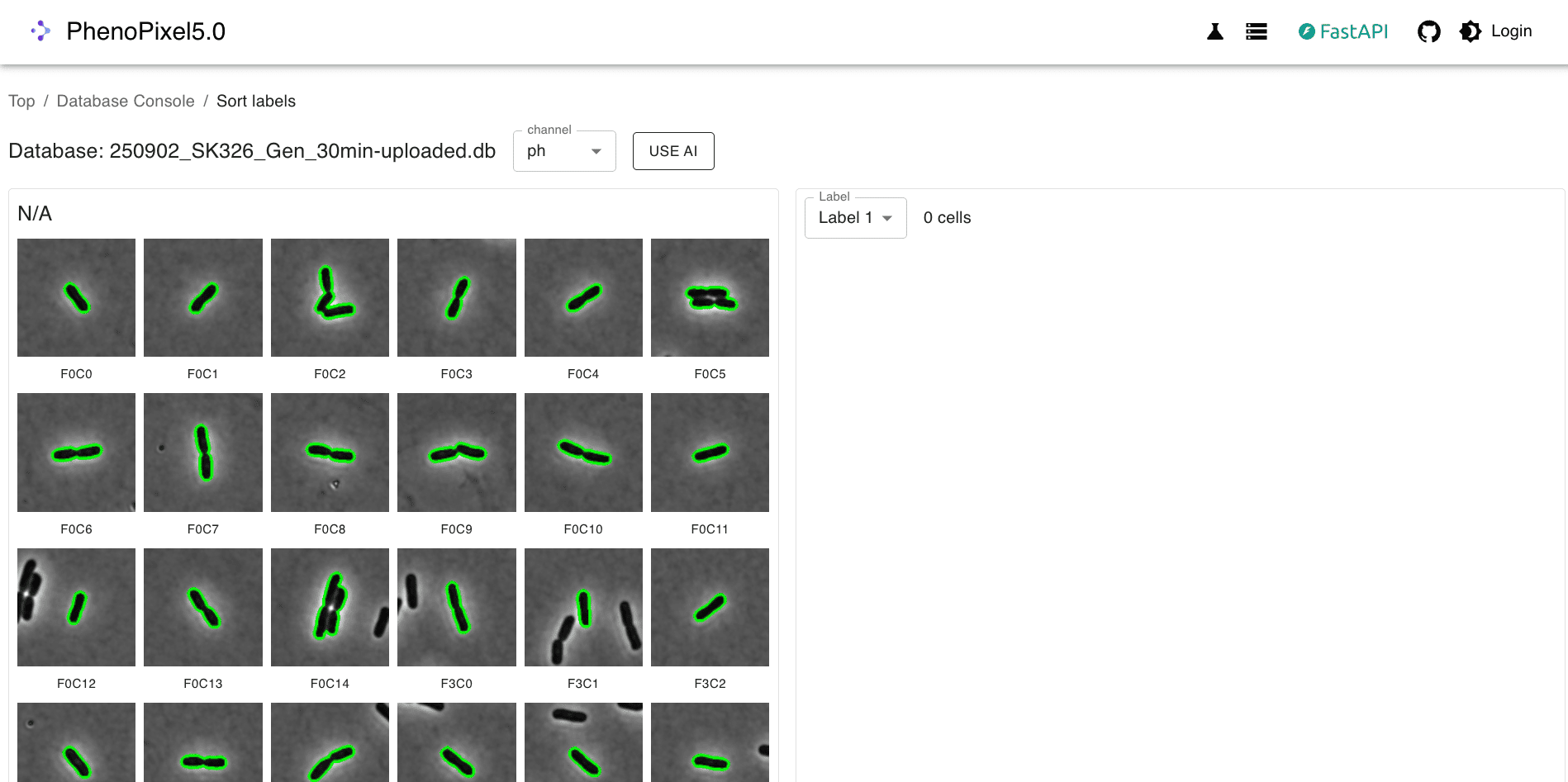
Sort Labels画面で、ラベルN/Aが「輪郭が整っていない細胞」、ラベル1が「正常に輪郭を取得できた細胞」となるように分類します。ラベル1に該当する細胞を選択してラベリングを進めます。
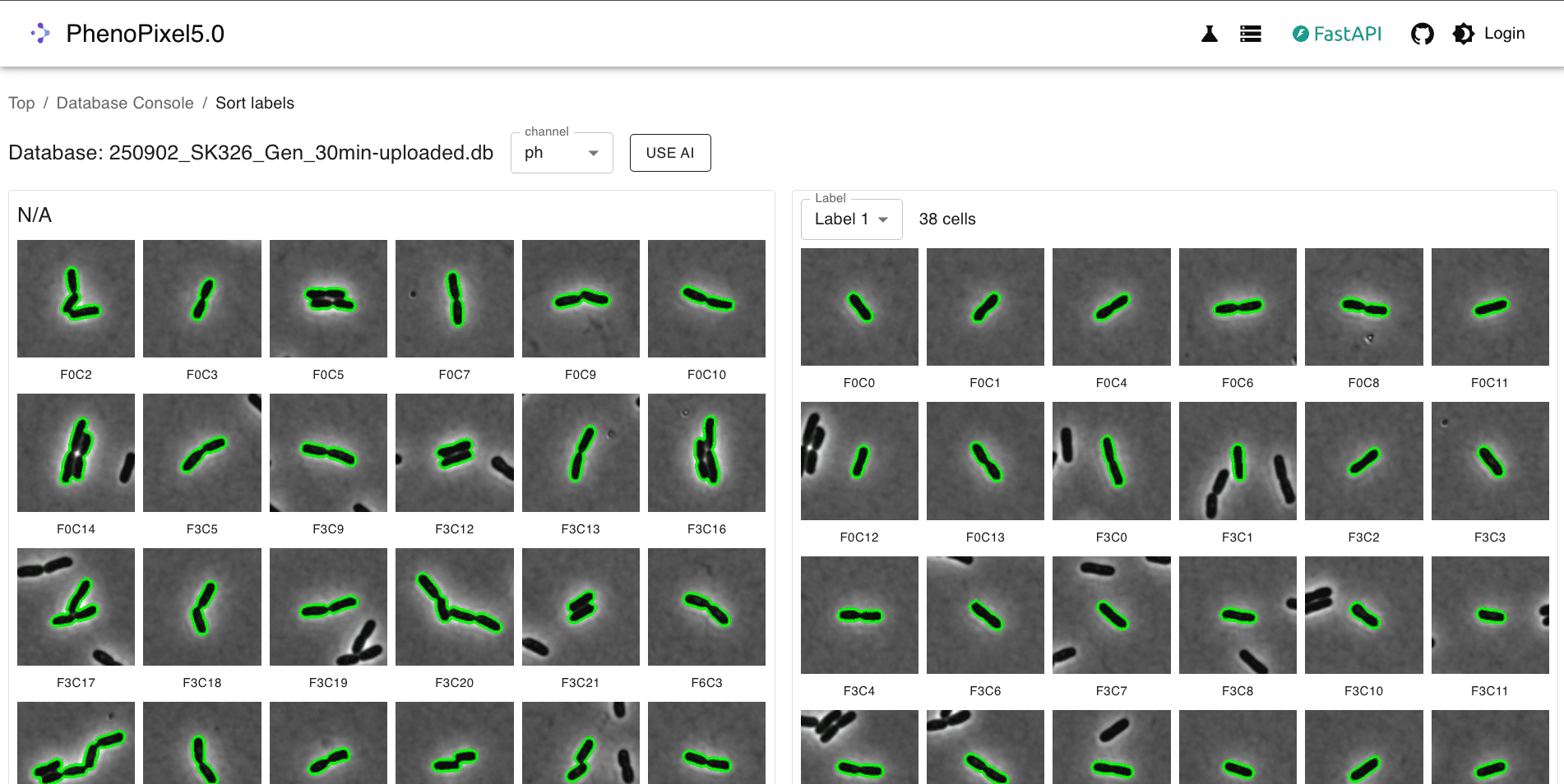
ラベリング終了後、画面右側に表示される集団がラベル1の細胞のみで構成されていることを確認します。
Database Consoleに戻り、mark as completeをクリックしてラベリングを完了します。
オープンソースで公開中。インストールやセットアップの手順は GitHub で詳しく確認できます。